|
|
| Line 1: |
Line 1: |
| - | ==Introducción a los modelos moleculares 3D== | + | =3D molecular models: an introduction= |
| | + | [[es:3D Molecular Models (spanish)]] |
| | [[Image:FormulasEmpiricas.png|300px|left|thumb|Formulas empíricas]] | | [[Image:FormulasEmpiricas.png|300px|left|thumb|Formulas empíricas]] |
| | [[Image:FormulasEstrucurales2.png|300px|left|thumb|Formulas Estructurales: moléculas pequeñas]] | | [[Image:FormulasEstrucurales2.png|300px|left|thumb|Formulas Estructurales: moléculas pequeñas]] |
| | [[Image:FormulasEstructurales.png|300px|left|thumb|Formulas Estructurales: compuestos orgánicos]] | | [[Image:FormulasEstructurales.png|300px|left|thumb|Formulas Estructurales: compuestos orgánicos]] |
| - | <big>Una de las actividades a las que necesariamente se han tenido que dedicar los científicos a lo largo de la historia es la que consiste en la elaboración de '''modelos''' que representen los fenómenos sobre los que trabajan de una manera que resulte accesible a la intuición humana. Esta actividad ha sido especialmente necesaria en el campo de la Química y, muy particularmente de la Bioquímica, ciencia en la que la representación de grandes estructuras moleculares, y de gran complejidad estructural, es totalmente imprescindible. | + | <big> |
| | + | <Structure load='' size='800' frame='true' align='right' caption='Modelos 3D' scene='60/603296/Agua1/1' /> |
| | | | |
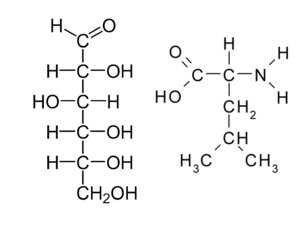
| - | :La forma más simple que se utilizado tradicionalmente para representar estructuras moleculares son las '''fórmulas químicas'''. Así, por medio de las llamadas '''fórmulas empíricas''', se representan las moléculas indicando los elementos químicos que las constituyen añadiendo a cada uno un subíndice que representa el número de átomos presentes de cada uno de ellos (ver figura a la izquierda del texto). Las fórmulas empíricas no dan información acerca de cómo están situados los átomos en la molécula ni de cómo están enlazados entre sí. Por ello, para conseguir una representación más próxima a la realidad, se han utilizado las llamadas '''fórmulas estructurales''', en las que se representan también por medio de sus símbolos los elementos químicos constituyentes y por medio de guiones los enlaces químicos que los unen. Las fórmulas estructurales resultan especialmente útiles en la Química Orgánica y en la Bioquímica, ciencias en las que la complejidad de las moléculas a representar es creciente. | + | :Small molecules (ball and stick models): |
| | + | ::<scene name='60/603296/Agua1/1'>Water</scene> |
| | + | ::<scene name='60/603296/Co2/1'>carbon dioxide</scene> |
| | + | ::<scene name='60/603296/Amoniaco/1'>Ammonia</scene> |
| | | | |
| - | :Aun siendo de gran utilidad, el uso de fórmulas, incluso de fórmulas estructurales, presenta la limitación de que con ellas se representa en dos dimensiones (sobre el plano del papel o pantalla) objetos tridimensionales como son las moléculas. Tradicionalmente se ha tratado de superar esta limitación utilizando convenciones en la representación que intentan dar al observador la sensación de profundidad o perspectiva (enlaces con trazo más o menos grueso, con forma de cuña, etc.). A pesar de ello, cuando se trata de moléculas muy grandes, las representaciones en dos dimensiones como las descritas van resultando cada vez más farragosas y menos útiles para proporcionarnos una idea cabal acerca de la forma tridimensional de la molécula representada. | + | :Glucose: |
| | + | ::<scene name='60/603296/Glucopiranosa/1'>Glucose</scene> (ball and stick model) |
| | + | ::<scene name='60/603296/Glucopiranosa/2'>Glucose</scene>(wireframe model) |
| | + | ::<scene name='60/603296/Glucopiranosa/3'>Glucose</scene>(spacefill model) |
| | + | ::<scene name='60/603296/Glucopiranosa/6'>Glucose</scene> (dots surface model) |
| | | | |
| - | :Para superar estas dificultades, al menos en el ámbito de la investigación científica, se ha recurrido a la construcción de '''modelos moleculares tridimensionales'''. Ensamblando convenientemente bolas y varillas fabricadas con los materiales adecuados se pueden construir representaciones a escala de todo tipo de moléculas. La construcción y manipulación de estos modelos moleculares resultó ser una herramienta fundamental en muchos de los avances en el estudio de la estructura de las macromoléculas que se produjeron en la segunda mitad del S.XX. Aun así, la construcción y manipulación de estos modelos suele ser un proceso lento y a veces tedioso para los investigadores. | + | :Macromolecules: |
| | + | ::<scene name='60/603296/Insulina/5'>Insulin</scene> (wireframe model) |
| | + | ::<scene name='60/603296/Insulina/7'>Insulin</scene> (ball and stick model) |
| | + | ::<scene name='60/603296/Insulina/8'>Insulin</scene> (dots surface model) |
| | + | ::<scene name='60/603296/Insulina/9'>Insulin</scene> (spacefill model) |
| | + | ::<scene name='60/603296/Insulina/20'>Ribbon model</scene> |
| | + | ::<scene name='60/603296/Insulina/22'>"Rockets model"</scene>. |
| | | | |
| - | ----
| + | :Color code (CPK) |
| | + | ::<scene name='60/603296/Insulina/10'>grey for carbon</scene> |
| | + | ::<scene name='60/603296/Insulina/11'>Red for oxygen</scene> |
| | + | ::<scene name='60/603296/Insulina/12'>Blue for nitrogen</scene> |
| | + | ::<scene name='60/603296/Insulina2/2'>Yellow for sulfur</scene> |
| | + | ::White for hydrogen |
| | | | |
| | + | :<scene name='60/603296/Insulina/7'>Other color codes</scene> |
| | + | ::<scene name='60/603296/Insulina/18'>Spectral color series</scene> |
| | + | ::<scene name='60/603296/Insulina/19'>Chain color</scene> |
| | | | |
| - | En la actualidad existen aplicaciones informáticas que permiten desarrollar y manipular virtualmente '''modelos moleculares tridimensionales''' sin los inconvenientes que suponen los modelos de bolas y varillas tradicionales. Además, se han desarrolado formas alternativas de representación, que permiten resaltar determinadas características de la molécula según las necesidades del usuario. En el recuadro de la derecha iremos haciendo un recorrido por distintos modelos 3D virtuales, desde los más simples a los de mayor complejidad. <Structure load='' size='800' frame='true' align='right' caption='Modelos 3D' scene='60/603296/Agua1/1' />
| + | :ATCase |
| - | | + | ::<scene name='60/603296/Atcasa/1'>Aspartate carbamoyltransferase</scene> |
| - | En primer lugar, vamos a ver algunos modelos de moléculas pequeñas como el <scene name='60/603296/Agua1/1'>agua</scene>, el <scene name='60/603296/Co2/1'>dióxido de carbono</scene>, o el <scene name='60/603296/Amoniaco/1'>amoníaco</scene>, representados aquí con modelos virtuales de '''bolas y varillas'''.
| + | ::<scene name='60/603296/Atcasa/2'>Catalitic trimers</scene> |
| - | | + | ::<scene name='60/603296/Atcasa/3'>Regulatory dimers</scene> |
| - | La gran utilidad de estos modelos se aprecia mejor cuando tenemos que representar moléculas algo más complejas como por ejemplo la <scene name='60/603296/Glucopiranosa/1'>glucosa</scene>, representada aquí en su forma cíclica como α-D-glucopiranosa en un modelo de bolas y varillas. Esta representación y su equivalente denominada <scene name='60/603296/Glucopiranosa/2'>"malla de alambre"</scene>(''wireframe''), resultan útiles para apreciar las distancias entre los átomos, los ángulos de enlace y otros aspectos de la geometría de la molécula.
| + | ::<scene name='60/603296/Atcasa/5'>Ribbon model</scene> |
| - | | + | ::<scene name='60/603296/Atcasa/4'>Ball and stick model</scene> |
| - | Si pretendemos formarnos una idea acerca de la forma de la superficie de la molécula, tal como sería "vista" por otras moléculas tales como enzimas, podemos recurrir a un <scene name='60/603296/Glucopiranosa/3'>modelo espacial compacto </scene>(''spacefill''), en el que los átomos se representa extendidos hasta su radio de Van der Waals, aunque de este modo nos perdemos algunos detalles de la geometría molecular. Existe una forma de representación alternativa que combina las ventajas de las dos anteriores: el modelo de <scene name='60/603296/Glucopiranosa/6'>superficie de puntos</scene>, en el que superpone un modelo de bolas y varillas con una "superficie transparente", perfilada a base de puntos.
| + | |
| - | | + | |
| - | Cuando se trata de representar macromoléculas como proteínas y ácidos nucleicos, los modelos moleculares virtuales resultan todavía de mayor utilidad. Veámoslo con una proteína de bajo peso molecular como la <scene name='60/603296/Insulina/5'>insulina</scene> humana, una hormona implicada en la regulación de metabolismo de los glúcidos, que está formada por dos cadenas polipeptídicas con un total de 51 residuos de aminoácidos. Aquí podemos apreciar la representación en '''malla de alambre'''. El <scene name='60/603296/Insulina/7'>modelo de bolas y varillas</scene> también resulta revelador de ciertos aspectos de la molécula. Si añadimos una <scene name='60/603296/Insulina/8'>superficie de puntos</scene> apreciamos ahora superpuesta una imagen de la superficie molecular de van der Waals, que se torna el aspecto más relevante de la representación en el <scene name='60/603296/Insulina/9'>modelo espacial compacto</scene>. En todas las representaciones hasta ahora expuestas se ha utilizado el código de colores CPK, propuesto inicialmente por los bioquímicos Corey, Pauling y Koltun y modificado posteriormente para adaptarlo a la generación de modelos moleculares virtuales. Este código asigna un color a cada uno de los elementos químicos más frecuentes atendiendo a la siguiente convención:<scene name='60/603296/Insulina/10'> gris para el carbono</scene>, <scene name='60/603296/Insulina/11'>rojo para el oxígeno</scene>, <scene name='60/603296/Insulina/12'>azul para el nitrógeno</scene> y <scene name='60/603296/Insulina2/2'>amarillo para el azufre</scene>. Se reserva el '''blanco''' para el '''hidrógeno''', que se obvia en la representación de macromoléculas complejas con el objeto de simplificarlas. Volvamos ahora al <scene name='60/603296/Insulina/7'>modelo de bolas y varillas</scene>. Además del código CPK que se ha descrito, se han desarrollado otras convenciones de colores para resaltar diferentes aspectos de la molécula a representar. Una de ellas, particularmente útil para las proteínas, consiste en la utilización de una <scene name='60/603296/Insulina/18'>gradación de colores</scene> que van variando desde el extremo amino-terminal (azul) hasta el carboxi-terminal (rojo) de una cadena polipeptídica. También existe la posibilidad de utilizar colores diferentes que pongan de manifiesto la existencia de <scene name='60/603296/Insulina/19'>varias cadenas polipeptídicas</scene> cuando es el caso.
| + | |
| - | | + | |
| - | En ocasiones puede resultar de interés que el modelo proporcione información acerca de la estructura secundaria que presentan distintos tramos de una cadena polipeptídica. Para ello se utiliza el <scene name='60/603296/Insulina/20'>modelo de cintas</scene>, que en este caso pone de manifiesto el predominio en la insulina de la estructura secundaria en hélice-α. Una alternativa interesante puede ser el modelo que resalta el <scene name='60/603296/Insulina/21'>esqueleto</scene> de las cadenas polipeptídicas obviando las cadenas laterales de los aminoácidos, o el que reprenta los tramos con estructura secundaria en hélice-α por medio de <scene name='60/603296/Insulina/22'>"cohetes"</scene>.
| + | |
| - | | + | |
| - | La molécula utilizada como ejemplo hasta ahora, la insulina, es una proteína relativamente pequeña y de estructura tridimensional no demasiado compleja. Los modelos moleculares virtuales pueden utilizarse para representar estructuras sensiblemente más grandes y complejas. Aquí podemos ver un modelo espacial compacto del enzima <scene name='60/603296/Atcasa/1'>aspartato-transcarbamilasa</scene>, que cataliza una de las etapas iniciales en la síntesis de las pirimidinas. La unidad funcional de la '''aspartato-transcarbamilasa''' es un ''dodecámero'' que aquí vemos representado mediante un modelo espacial compacto en el que se resaltan por medio de distintos colores las 12 subunidades que lo forman. En el dodecámero podemos distinguir <scene name='60/603296/Atcasa/2'>dos trímeros catalíticos</scene> y <scene name='60/603296/Atcasa/3'>tres dímeros reguladores</scene>. Un <scene name='60/603296/Atcasa/5'>modelo de cintas</scene> nos da información acerca de la distribución de los distintos tipos de estructura secundaria en las distintas subunidades; vemos que la hélice-α (en púrpura) predomina en las subunidades catalíticas mientras que la hoja-β (en amarillo) lo hace en las reguladoras. Usando ahora un <scene name='60/603296/Atcasa/4'>modelo de bolas y varillas</scene> podemos apreciar la compleja estructura de esta gran macromolécula que está formada por 21.318 átomos.
| + | |
| | | | |
| | == Atribución de contenidos == | | == Atribución de contenidos == |
| - | *Los modelos moleculares de agua, dióxido de carbono, amoníaco y α-D-glucopiranosa han sido generados por el autor utilizando la herramienta JSME Molecular Editor [http://biomodel.uah.es/en/DIY/JSME/draw.es.htm] | + | *Water, carbon dioxide and ammonia molecular model were generated using tool JSME Molecular Editor [http://biomodel.uah.es/en/DIY/JSME/draw.es.htm] |
| - | *El modelo molecular de la ''insulina'' se ha tomado de Proteopedia [[3i40]] | + | *Insulin molecular models are based on Proteopedia file [[3i40]] |
| - | *El modelo molecular del enzima ''aspartato transcarbamilasa'' se ha tomado de [[Aspartate Transcarbamoylase (ATCase)]] | + | *ATCase molecular models are based on Proteopedia file [[Aspartate Transcarbamoylase (ATCase)]] |
| - | | + | |
| | <references/> | | <references/> |