Beta-galactosidase (hebrew)
From Proteopedia
(Difference between revisions)
| Line 88: | Line 88: | ||
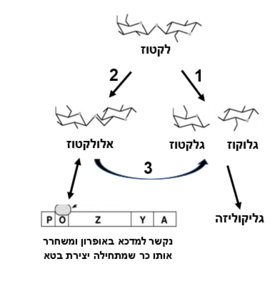
ישנו אזור בכל מונומר הקריטי לחיבורם לכדי טטרמר פעיל. כאשר אזור זה חסר, מתקבל אנזים לא פעיל. | ישנו אזור בכל מונומר הקריטי לחיבורם לכדי טטרמר פעיל. כאשר אזור זה חסר, מתקבל אנזים לא פעיל. | ||
<br> | <br> | ||
| - | הוספת המקטע החסר, תרכיב מחדש את האנזים הפעיל. הוספה כזו נקראת השלמת אלפא(ראה | + | הוספת המקטע החסר, תרכיב מחדש את האנזים הפעיל. הוספה כזו נקראת השלמת אלפא (ראה פסקה שימוש במחקר והנדסה גנטית). |
<br> | <br> | ||
| - | פעילות האנזים תלויה גם בקשירת נתרן ואשלגן בעמדות מסוימות של החלבון (עמדות אלה שמורות באבולוציה, ז"א מופיעות בבטא גלקטוזידאז של אורגניזמים שונים). | + | פעילות האנזים תלויה גם בקשירת נתרן ואשלגן בעמדות מסוימות של החלבון (עמדות אלה שמורות באבולוציה, ז"א מופיעות בבטא גלקטוזידאז של אורגניזמים שונים. ראה פסקה הבאה). |
<br> | <br> | ||
| - | ישנם מספר <scene name='89/898980/Mg_and_na/1'>יוני נתרן ומגנזיום</scene> הקשורים לחומצות אמיניות שונות בחלבון. תפקידם ככל הנראה לאפשר ייצוב של המבנה המרחבי של כל אחד משני הדימרים המרכיבים את הטטרמר | + | ישנם מספר <scene name='89/898980/Mg_and_na/1'>יוני נתרן ומגנזיום</scene> הקשורים לחומצות אמיניות שונות בחלבון. תפקידם ככל הנראה לאפשר ייצוב של המבנה המרחבי של כל אחד משני הדימרים המרכיבים את הטטרמר, |
<br> | <br> | ||
| - | ביניהם יון מגנזיום הקשור באתר הפעיל מאפשר פעילות מיטבית של האנזים | + | ביניהם <scene name='89/898980/Active_site_mg/1'>יון מגנזיום הקשור באתר הפעיל</scene> מאפשר פעילות מיטבית של האנזים. |
| Line 114: | Line 114: | ||
<scene name='89/898980/Mg_and_na/1'>יוני נתרן ומגנזיום</scene> | <scene name='89/898980/Mg_and_na/1'>יוני נתרן ומגנזיום</scene> | ||
<br> | <br> | ||
| - | <scene name='89/898980/Active_site_mg/1'>מגנזיום באתר הפעיל</scene> | + | <scene name='89/898980/Active_site_mg/1'>יון מגנזיום הקשור באתר הפעיל</scene> |
This is a sample scene created with SAT to <scene name="/12/3456/Sample/1">color</scene> by Group, and another to make <scene name="/12/3456/Sample/2">a transparent representation</scene> of the protein. You can make your own scenes on SAT starting from scratch or loading and editing one of these sample scenes. | This is a sample scene created with SAT to <scene name="/12/3456/Sample/1">color</scene> by Group, and another to make <scene name="/12/3456/Sample/2">a transparent representation</scene> of the protein. You can make your own scenes on SAT starting from scratch or loading and editing one of these sample scenes. | ||
Revision as of 08:56, 4 January 2022
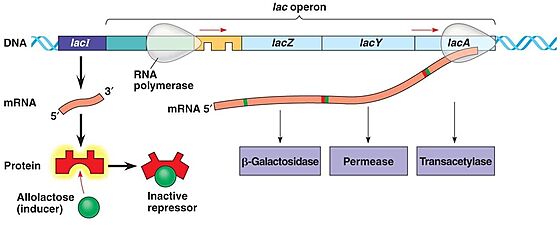
בטא-גלקטוזידאז
| |||||||||||
References
- ↑ Hanson, R. M., Prilusky, J., Renjian, Z., Nakane, T. and Sussman, J. L. (2013), JSmol and the Next-Generation Web-Based Representation of 3D Molecular Structure as Applied to Proteopedia. Isr. J. Chem., 53:207-216. doi:http://dx.doi.org/10.1002/ijch.201300024
- ↑ Herraez A. Biomolecules in the computer: Jmol to the rescue. Biochem Mol Biol Educ. 2006 Jul;34(4):255-61. doi: 10.1002/bmb.2006.494034042644. PMID:21638687 doi:10.1002/bmb.2006.494034042644