DD-transpeptidase (Hebrew)
From Proteopedia
| Line 4: | Line 4: | ||
</p> | </p> | ||
===<div style="text-align:right;direction:rtl;">'''מבנה החלבון'''</div>=== | ===<div style="text-align:right;direction:rtl;">'''מבנה החלבון'''</div>=== | ||
| + | <p dir='rtl'> | ||
| + | גודל [https://www.youtube.com/watch?v=aFwOfiPlSdc האנזים] טרנספפטידאז הוא 37.5 קילו דלתון והוא בנוי מ406 [https://www.youtube.com/watch?v=gpu2AUW4S5g&ab_channel=ComGeek חומצות אמינו]. בדגם שגובש ומוצג בערך זה יש את <scene name='89/898977/Amino_acids/2'>347 חומצות האמינו</scene> המייצגות את החלבון הפעיל (עמדות 32-380) (4). המקטע שלא גובש כולל שרשרת פפטידית של 31 חומצות אמינו (1-31) הנחתכת מהחלבון לאחר שהוא מגיע ליעדו בדופן התא, ועוד מקטע של 26 חומצות אמינו (381-406) הנחתך מהחלבון לאחר אקטיבציה(3). 60% מהחלבון מורכב משרשראות פוליפפטידיות בעלות מבנה שניוני (סלילי אלפא ומשטחי בטא). האתר הפעיל של האנזים נוצר ע"י 4 חומצות אמינו בסיסיות ועוד 4 חומצות אמינו ארומטיות כאשר סרין במיקום 62 היא החומצה האמינית אשר קושרת אליה את הסובסטרט (1). | ||
| + | </p> | ||
| + | <br> | ||
| + | <p dir='rtl'> | ||
| + | באתר הפעיל יש 8 חומצות אמיניות הנמצאו שמורות אבולוציונית בכל סוגי האנזימים ממשפחת הפפטידאזות. חומצות אמינו אלו ממוקמות בשלושה אזורים שונים באתר הפעיל והן יוצרות את הכיס הקטליטי של האנזים. סרין62 וליזין65 אחראיות לתגובה הכימית עם הסובסטראט(2), ושתי הקבוצות: טירוזין159-סרין160-אספרגין161, היסטידין298-תראונין299-גליצין300, הן חלק מאתרי הקישור לסובסטרט. | ||
| + | </p> | ||
| + | |||
<StructureSection load='3PTE' size='340' side='right' caption='Caption for this structure' scene=''> | <StructureSection load='3PTE' size='340' side='right' caption='Caption for this structure' scene=''> | ||
This is a default text for your page '''DD-transpeptidase (Hebrew)'''. Click above on '''edit this page''' to modify. Be careful with the < and > signs. | This is a default text for your page '''DD-transpeptidase (Hebrew)'''. Click above on '''edit this page''' to modify. Be careful with the < and > signs. | ||
You may include any references to papers as in: the use of JSmol in Proteopedia <ref>DOI 10.1002/ijch.201300024</ref> or to the article describing Jmol <ref>PMID:21638687</ref> to the rescue. | You may include any references to papers as in: the use of JSmol in Proteopedia <ref>DOI 10.1002/ijch.201300024</ref> or to the article describing Jmol <ref>PMID:21638687</ref> to the rescue. | ||
| - | <scene name='89/898977/Amino_acids/2'>חומצות האמינו | + | <scene name='89/898977/Amino_acids/2'>347 חומצות האמינו</scene> |
<br> | <br> | ||
<scene name='89/898977/Secondary_structures/1'>מבנה שניוני</scene> | <scene name='89/898977/Secondary_structures/1'>מבנה שניוני</scene> | ||
Revision as of 09:29, 4 January 2022
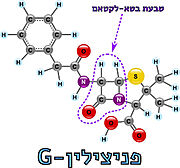
טרנספפטידאז
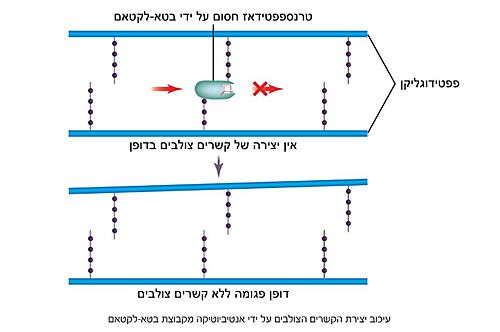
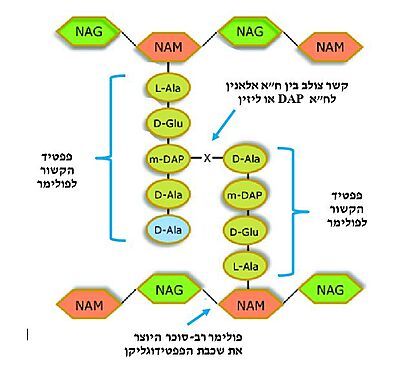
טרנספפטידאז (D-alanyl-D-alanine Peptidase) הוא אנזים חיידקי המעורב בתהליך הסינתזה של דופן התא בחיידקים. הסובסטראט של אנזים זה היא דופן תא החיידק, הבנויה משרשראות ארוכות של פפטידוגליקן המחוברות זו לזו בקשר פפטידי ליצירת מבנה יציב וקשיח. אנטיביוטיקה מסוג בטא-לקטאם (לדוגמה: פניצילין) מעכבת באופן תחרותי ובלתי הפיך את האנזים, כך שנוצרת דופן פגומה, החיידק אינו עמיד בלחצים אוסמוטיים וכתוצאה מכך התא מתפוצץ והחיידק מת.
מבנה החלבון
גודל האנזים טרנספפטידאז הוא 37.5 קילו דלתון והוא בנוי מ406 חומצות אמינו. בדגם שגובש ומוצג בערך זה יש את המייצגות את החלבון הפעיל (עמדות 32-380) (4). המקטע שלא גובש כולל שרשרת פפטידית של 31 חומצות אמינו (1-31) הנחתכת מהחלבון לאחר שהוא מגיע ליעדו בדופן התא, ועוד מקטע של 26 חומצות אמינו (381-406) הנחתך מהחלבון לאחר אקטיבציה(3). 60% מהחלבון מורכב משרשראות פוליפפטידיות בעלות מבנה שניוני (סלילי אלפא ומשטחי בטא). האתר הפעיל של האנזים נוצר ע"י 4 חומצות אמינו בסיסיות ועוד 4 חומצות אמינו ארומטיות כאשר סרין במיקום 62 היא החומצה האמינית אשר קושרת אליה את הסובסטרט (1).
באתר הפעיל יש 8 חומצות אמיניות הנמצאו שמורות אבולוציונית בכל סוגי האנזימים ממשפחת הפפטידאזות. חומצות אמינו אלו ממוקמות בשלושה אזורים שונים באתר הפעיל והן יוצרות את הכיס הקטליטי של האנזים. סרין62 וליזין65 אחראיות לתגובה הכימית עם הסובסטראט(2), ושתי הקבוצות: טירוזין159-סרין160-אספרגין161, היסטידין298-תראונין299-גליצין300, הן חלק מאתרי הקישור לסובסטרט.
| |||||||||||
References
- ↑ Hanson, R. M., Prilusky, J., Renjian, Z., Nakane, T. and Sussman, J. L. (2013), JSmol and the Next-Generation Web-Based Representation of 3D Molecular Structure as Applied to Proteopedia. Isr. J. Chem., 53:207-216. doi:http://dx.doi.org/10.1002/ijch.201300024
- ↑ Herraez A. Biomolecules in the computer: Jmol to the rescue. Biochem Mol Biol Educ. 2006 Jul;34(4):255-61. doi: 10.1002/bmb.2006.494034042644. PMID:21638687 doi:10.1002/bmb.2006.494034042644