Human Insulin Degrading Enzyme (Hebrew)
From Proteopedia
| Line 12: | Line 12: | ||
<p dir='rtl'> | <p dir='rtl'> | ||
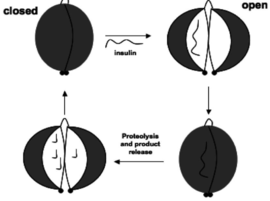
| - | חלבון זה הוא למעשה דימר, המורכב משני מונומרים (מוצגות בתכלת ובורוד) | + | חלבון זה הוא למעשה הומו-דימר, המורכב משני מונומרים (מוצגות בתכלת ובורוד). כדימר, שני המונומרים יכולים להיקשר זה לזה בקצוות שלהם ולשנות את הקונפורמציה המרחבית של האנזים (מפתוחה ולסגורה ולהפך, כפי שיתואר בהמשך). |
<br> | <br> | ||
| Line 33: | Line 33: | ||
<br> | <br> | ||
<br> | <br> | ||
| - | + | האנזים מפרק אינסולין נחשב למטאלופרוטאז, בהיותו המזרז את ריאקציית [https://www.youtube.com/watch?v=H9VTB41jgLI ההידרוליזה] של פפטידים באמצעות שימוש ביוני אבץ. ישנן שלוש <scene name='84/846755/Metal_binding_sites/2'>עמדות קישור</scene> המסומנות בבורדו (108 ,112 ,190) ליון המתכת אבץ Zn+2, משמע ששלושת האתרים הללו משתתפים יחד בקשירה של יון אבץ אחד לכל מונומר, ויחד הם מהווים חלק מהאתר הפעיל הפרוטאוליטי בכל אחד מהמונומרים המרכיבים את הדימר. בכל העמדות הללו מופיעה החומצה האמינית היסטידין (H)המכילה בשייר שלה קבוצה המכונה [https://en.wikipedia.org/wiki/Imidazole אימידזול] אשרה יכולה לקשור יוני מתכת (כדוגמת אבץ וניקל) בקשרים לא קוולנטים. עמדות אלו שמורות מבחינה אבולוציונית. | |
בסמיכות רבה לעמדות קושרות האבץ נמצא האתר הפעיל בעמדה 111, שם מתבצע הביקוע של הסובסטרט. | בסמיכות רבה לעמדות קושרות האבץ נמצא האתר הפעיל בעמדה 111, שם מתבצע הביקוע של הסובסטרט. | ||
למרות שרב הנסתר על הגלוי באשר למכניזם שמסביר את הקטליזה אותה מבצע האנזים, נמצא כי [https://he.wikipedia.org/wiki/%D7%A7%D7%99%D7%A0%D7%98%D7%99%D7%A7%D7%94_%D7%90%D7%A0%D7%96%D7%99%D7%9E%D7%98%D7%99%D7%AA שלב קובע מהירות] מותנה [https://he.wikipedia.org/wiki/%D7%94%D7%AA%D7%9E%D7%A8%D7%94_%D7%A0%D7%95%D7%A7%D7%9C%D7%90%D7%95%D7%A4%D7%99%D7%9C%D7%99%D7%AA התקפה נוקלאופילית] שמתרחשת כאשר יוני האבץ עוברים לסובסטרט. | למרות שרב הנסתר על הגלוי באשר למכניזם שמסביר את הקטליזה אותה מבצע האנזים, נמצא כי [https://he.wikipedia.org/wiki/%D7%A7%D7%99%D7%A0%D7%98%D7%99%D7%A7%D7%94_%D7%90%D7%A0%D7%96%D7%99%D7%9E%D7%98%D7%99%D7%AA שלב קובע מהירות] מותנה [https://he.wikipedia.org/wiki/%D7%94%D7%AA%D7%9E%D7%A8%D7%94_%D7%A0%D7%95%D7%A7%D7%9C%D7%90%D7%95%D7%A4%D7%99%D7%9C%D7%99%D7%AA התקפה נוקלאופילית] שמתרחשת כאשר יוני האבץ עוברים לסובסטרט. | ||
Revision as of 07:04, 16 July 2020
| |||||||||||
References
1. Affholter JA, Fried VA, Roth RA (December 1988). "Human insulin-degrading enzyme shares structural and functional homologies with E. coli protease III". Science. 242 (4884): 1415–8.
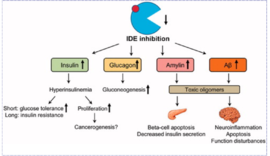
2. Farris W, Mansourian S, Chang Y, Lindsley L, Eckman EA, Frosch MP, et al. (April 2003). "Insulin-degrading enzyme regulates the levels of insulin, amyloid beta-protein, and the beta-amyloid precursor protein intracellular domain in vivo". Proceedings of the National Academy of Sciences of the United States of America. 100 (7): 4162–7.
3. Hulse RE, Ralant LA, Tang WJ (February 2009). "Structure, Function, and Regulation of Insulin-Degrading Enzyme". Vitamins & Hormones 80:635-48. doi: 10.1016/S0083-6729(08)00622-5
4. Kerr ML, Small DH (April 2005). "Cytoplasmic domain of the beta-amyloid protein precursor of Alzheimer's disease: function, regulation of proteolysis, and implications for drug development". Journal of Neuroscience Research. 80 (2): 151–9. doi:10.1002/jnr.20408. PMID 15672415.
5. Kurochkin IV, Goto S (May 1994). "Alzheimer's beta-amyloid peptide specifically interacts with and is degraded by insulin degrading enzyme". FEBS Letters. 345 (1): 33–7. doi:10.1016/0014-5793(94)00387-4. PMID 8194595.
6. Mirsky IA, Broh-Kahn RH (January 1949). "The inactivation of insulin by tissue extracts; the distribution and properties of insulin inactivating extracts". Archives of Biochemistry. 20 (1): 1–9. PMID 18104389.
7.Wang DS, Dickson DW, Malter JS (2006). "beta-Amyloid degradation and Alzheimer's disease". Journal of Biomedicine & Biotechnology. 2006 (3): 58406. doi:10.1155/JBB/2006/58406. PMC 1559921. PMID 17047308.
8. https://www.ncbi.nlm.nih.gov/sites/entrez?Db=gene&Cmd=ShowDetailView&TermToSearch=3416
9. https://www.uniprot.org/uniprot/P14735
10. https://www.rcsb.org/structure/6b7y