This is a default text for your page Beta-galactosidase (hebrew). Click above on edit this page to modify. Be careful with the < and > signs.
You may include any references to papers as in: the use of JSmol in Proteopedia [1] or to the article describing Jmol [2] to the rescue.
כללי
בטא גלקטוזידאז הוא אנזים הקיים בחיידקי E.coli, ובאורגניזמים רבים אחרים, האחראי על פירוק לקטוז (דו סוכר המורכב מגלוקוז ופרוקטוז).
חד סוכרים אלו מהווים מקורות אנרגיה לחיידק (החיידק מפרק אותם בתהליך
גליקוליזה)
.בחיידקי E.coli, האנזים הוא תוצר של הגן lacZ
שהינו חלק מאופרון הלקטוז. רצפו וארגון הגנים באופרון התגלו ב-1961.
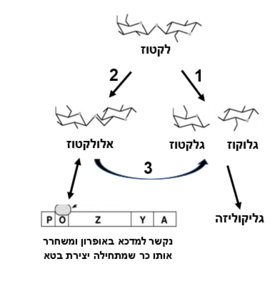
האנזים בטא גלקטוזידאז מבצע 3 פעולות שמביאות בסופו של דבר לפירוק הלקטוז לגלוקוז וגלקטוז:
- ביקוע הלקטוז לגלקטוז+גלוקוז
- המרת הלקטוז לאלולקטוז
- הידרוליזה לאלולקטוז לגלקטוז+גלוקוז
לתוצר אלולקטוז יש תפקיד בבקרה הגנטית על בטאגלקטוזידאז (ראה פסקה על אופרון הלקטוז)
הגן המקודד לאנזים בטא גלקטוזידאז נמצא גם בגנום של בני אדם (הרבה פעמים האנזים המקודד על ידי גן זה מכונה: לקטאז).
גן זה מתבטא בעיקר בתקופת הינקות. אצל רוב בני האדם (כ-70%) פעילות האנזים הולכת ופוחתת לאחר תקופה זו כיוון שרמת הביטוי שלו יורדת,
מה שגורם להרבה אנשים לבטא אי סבילות ללקטוז (כיוון שהלקטוז לא מתפרק).
חברות מזון שונות מוסיפות את האנזים, המופק מאורגנזימים שונים, למזונות המכילים לקטוז כדי שהאנזים יפרק את הלקטוז
וכך גם אנשים בעלי אי סבילות ללקטוז יכולים להנות ממזונות אלה.
לאחרונה נחקר האנזים כטיפול אפשרי לאי-סבילות ללקטוז על ידי ריפוי גני, בהחדרת הגן לאנזים זה ל-DNA, על מנת שאנשים החסרים לקטאז יוכלו לבקע לקטוז בדרך זו.
אופרון הלקטוז
מאחר ולקטוז הוא לא מקור האנרגיה המועדף על החיידק E.coli הוא לא מנצל אותו כמקור אנרגיה בתנאים בהם קיימים בסביבתו מקורות אנרגיה עדיפים היות ותהליך יצירת החלבונים הוא תהליך צורך אנרגיה, על החיידק לבטא את החלבונים המפרקים לקטוז רק כאשר הלקטוז משמש כמקור ההזנה שלו. בהיעדר לקטוז, תופעל בקרה גנטית אשר תמנע ייצור מיותר של האנזים.
מבנה אופרון הלקטוז:
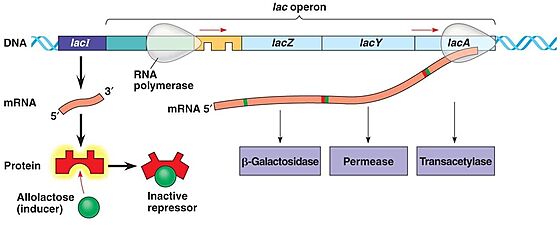
אופרון הלקטוז כולל 3 גנים מבניים, ושני רצפי DNA בקרתיים : פרומוטור ואופרטור (כמפורט מטה). בנוסף, הבקרה על שעתוק האופרון מצריכה מעורבות של חלבון הקרוי רפרסור.
חלבון זה מבוטא מגן הקרוי LacI המצוי בגנום בסמיכות לאופרון הלקטוז.
Lac Z- גן שמקודד ל-β-galactosidase – אנזים שמפרק לקטוז לגלוקוז ולגלקטוז
Lac Y- גן שמקודד ל-β-galactosidase permease, נשא שמאפשר ללקטוז להיכנס אל התא.
Lac A- גן שמקודד לאנזים טראנסאצטילאז שמסייע בניצול הלקטוז.
פרומוטור: מקטע דנ"א שנמצא לפני האופרון ואחראי על גיוס ה-RNA polymerase לשעתוק הגנים המבניים.
Lac I- גן המקודד לחלבון רפרסור, דכאן.
רפרסור (דכאן): חלבון שהקשירה שלו לאופרטור מעכבת את הביטוי של הגן.
אופרטור: אתר קשירה לחלבון הרפרסור (דכאן)
אינדוסר (משרן): מולקולת אלולקטוז שהיא אחד מתוצרי הפירוק של הלקטוז, משמשת כמשרן בתהליך.
בקרה שלילית באופרון הלקטוז-
כאשר חלבון הרפרסור נקשר לאתר האופרטור, הוא מונע את קשירתו של האנזים RNA polymerase לאתר הפרומוטור וכך נמנע תעתוק האופרון.
אלולקטוז, תוצר הפירוק של לקטוז, נקשר לחלבון הרפרסור, משנה את מבנהו המרחבי ומסיר אותו למהאופרטור. זה מאפשר את קשירת RNA polymerase לפרומוטור ומתבצע תעתוק של האופרון.
מבנה הבטא גלקטוזידאז
החלבון בטא גלאקטוזידאז מהאורגניזם E.coli הינו טטרמר שמורכב מ,כל מונומר מורכב מ- 1023
ישנם ארבעה אתרים פעילים, בכל מונומר.
האתרים הפעילים פועלים באופן עצמאי, אך פעילותם תלויה במבנה התקין של הטטרמר.
ישנו אזור בכל מונומר הקריטי לחיבורם לכדי טטרמר פעיל. כאשר אזור זה חסר, מתקבל אנזים לא פעיל.
הוספת המקטע החסר, תרכיב מחדש את האנזים הפעיל. הוספה כזו נקראת השלמת אלפא (ראה פסקה שימוש במחקר והנדסה גנטית).
פעילות האנזים תלויה גם בקשירת נתרן ואשלגן בעמדות מסוימות של החלבון (עמדות אלה שמורות באבולוציה, ז"א מופיעות בבטא גלקטוזידאז של אורגניזמים שונים. ראה פסקה הבאה).
ישנם מספר הקשורים לחומצות אמיניות שונות בחלבון. תפקידם ככל הנראה לאפשר ייצוב של המבנה המרחבי של כל אחד משני הדימרים המרכיבים את הטטרמר,
ביניהם מאפשר פעילות מיטבית של האנזים.
בטא-גלקטוזידאז בראי האבולוציה
דמיון ברצפי DNA וחלבונים לאורך האבולוציה מעיד על חשיבות החלבון (או איזור בחלבון) לתפקוד האורגניזמים.
הצטברות מוטציות לאורך האבולוציה הביאה לשינויים גנטיים מהותיים אשר איפשרו יצירה של מגוון רחב של יצורים, השונים גנטית זה מזה.
אזורים בעלי חשיבות פחותה לתפקוד חלבון מסוים מציגים שינויים גנטיים גדולים יותר בין אורגניזם אחד לאחר.
עם זאת, בחלבונים שונים ניתן לראות עמדות ששמורות לאורך האבולוציה, גם בין יצורים רחוקים אבולוציונית זה מזה.
העובדה שאותן עמדות שמורות ביצורים שרחוקים אבולוציונית זה מזה, מעידה על חשיבות העמדות אלו לתפקוד החלבון.
במבנה החלבוני של האנזים בטא-גלקטוזידאז יש מספר אתרים החשובים לתפקודו.
בהשוואת רצפים של החלבון מאורגניזמים שונים מצביעה על מספר .
לדוגמא: חומצה אמינית בעמדות 462 ו- 538 שמהווה את .
ניתן להשוות עמדות שמורות באבולוציה באמצעות עימוד רצפים בכלים ביואינפורמטים שונים כמו
clustalw. כל פסקה מציגה עימוד בין 9 אורגניזמים נבחרים. שם האורגניזם כתוב בעמודה השלישית משמאל, ואחריו רצף חומצות האמיניות של אותו אורגניזם בחלבון מסוים.
עמדות שמורות בכל האורגניזמים שנבדקו: מסומנות ב- *
עמדות שמורות ברוב האורגניזמים אך לא בכולם: מסומנות ב- :
עמדות לא שמורות: ללא סימון
הדוגמא מציגה עימוד של האנזים בטא גלקטוזידאז בחיידקים שונים, בהשוואה לחיידק
E.coli, אשר נמצא בשורה הראשונה. עמדות שמורות המסומנות באדום הן עמדות חשובות בתפקוד החלבון.

תוצאות עימוד בטא-גלקטוזידאז
שימוש במחקר והנדסה גנטית
שתי תגליות בנוגע לאנזים אפשרו את השימוש בו לצרכי מחקר:
1.האנזים מאוד ספציפי לגלקטוז אך פחות לגלוקוז, ולכן אפשר לגדל תאים בנוכחות גלקטוז שמחובר לחומר אחר, כך שנוצר חומר שהוא אנלוג ללקטוז והאנזים עדיין יפרק אותו. אחד מהחומרים האלה, X-gal, מכיל צבע שהופך לכחול כשהוא מופרד מהגלקטוז.
2.השלמת אלפא- ניתן להשבית את פעילות האנזים על ידי הסרת פפטיד מסוים (פפטיד אלפא) מהמבנה השלם ולהפעילו מחדש על ידי שילוב הפפטיד שנשאר (פפטיד אומגה) עם פפטידים משלימים שכוללים את פפטיד אלפא.
המדענים משתמשים בשתי התגליות הללו כדי לעקוב אחר תהליכים המתרחשים בחיידקים מהונדסים:
שימוש כגן מבחין
שיטת מעקב זו הגן לאנזים משמש כגן מבחין המאפשר להבדיל בין פלסמידים שקלטו מחדר בתהליך השיבוט לפלסמידים שלא קלטו אותו.
השיטה: מכינים חיידקים ללא עמידות לאנטיביוטיקה שהגנום שלהם מכיל גן מוטנטי, המקודד לפפטיד אומגה בלבד (שאינו פעיל ללא פפטיד אלפא).
מכינים פלסמיד שהוחדר אליו גן לעמידות לאנטיביוטיקה באזור אחד וגן לפפטיד אלפא (שאינו פעיל ללא פפטיד אומגה) באזור אחר. לתוך אזור הגן לפפטיד אלפא מחדירים את המקטע שאותו רוצים לשבט, כך שהוא "מפריע" לתעתוק הגן לפפטיד אלפא (ראה איור).
מחדירים את הפלסמידים לחיידקים שהוכנו (חסרי הגן לפפטיד אלפא) ואת החיידקים זורעים על מצע שמכיל את ה X-gal ואת האנטיביוטיקה הרלוונטית.
Structural highlights
This is a sample scene created with SAT to by Group, and another to make of the protein. You can make your own scenes on SAT starting from scratch or loading and editing one of these sample scenes.