User:Clara Fernández Vidal/Isocitrato deshidrogenasa
From Proteopedia
| Line 34: | Line 34: | ||
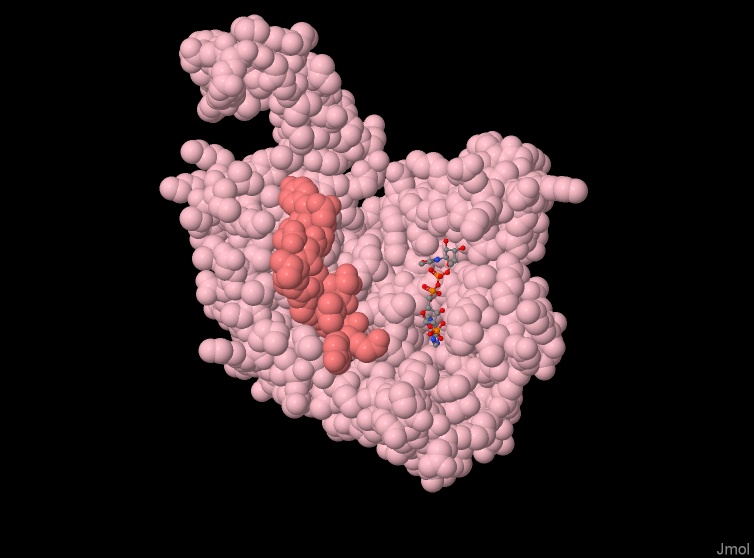
La conformación de la subunidad A (rojo) en el complejo binario (IDH-NADP) se parece más a la forma abierta. Proponen que la conformación abierta de IDH puede representar a la forma inactiva de la enzima. | La conformación de la subunidad A (rojo) en el complejo binario (IDH-NADP) se parece más a la forma abierta. Proponen que la conformación abierta de IDH puede representar a la forma inactiva de la enzima. | ||
La conformación de IDH de la subunidad B (azul) del complejo binario (IDH-NADP) está entre la forma abierta y la cerrada. Proponen que la forma semiabierta de la IDH puede representar una forma intermedia entre la conformación activa y la inactiva. | La conformación de IDH de la subunidad B (azul) del complejo binario (IDH-NADP) está entre la forma abierta y la cerrada. Proponen que la forma semiabierta de la IDH puede representar una forma intermedia entre la conformación activa y la inactiva. | ||
| + | |||
| + | '''Escena 0. Conservación evolutiva" | ||
| + | <scene name='10/1037483/Conservacion_dimero_1t09/2'>Escena 2</scene> | ||
| + | |||
'''Escena 2. Superposición de las subunidades A y B de IT09''' | '''Escena 2. Superposición de las subunidades A y B de IT09''' | ||
Revision as of 10:02, 18 April 2024
Isocitrato deshidrogenasa: escenas 3D para la comprensión de su función y estructura
El objetivo de la publicación de este artículo fue enriquecer con escenas 3D la literatura existente sobre la conocida enzima Isocitrato deshidrogenasa (IDH) de manera que se pudieran observar mejor las interacciones de los ligandos con la enzima, así como entender los cambios conformacionales que causan las mutaciones en los centros activos de esta. En otras palabras, se demostró con la visualización 3D una mejora en el estudio de estructura y función de biomoléculas.
Criterios de búsqueda en Protein Data Bank (PDB)
Se procedió a estudiar la Isocitrato deshidrogenasa (IDH) a partir de literatura existente. Primeramente, se hizo una búsqueda en PDB [1] de Isocitrate dehydrogenase y se filtró para seleccionar solo aquellas enzimas que fueran pertenecientes a Homo sapiens, aparecieron 16 resultados.
Los códigos de PBD de las enzimas con las que se trabajó fueron: 1T09,1T0L, 3MAP y 3MAS por la accesibilidad a la literatura y el interés de su contenido
La enzima y sus isoformas
La IDH es una importante enzima en el metabolismo celular humano formando parte del ciclo de Krebs, catalizando la descarboxilación oxidativa del Isocitrato (ICT) en α-cetoglutarato (αKG). Las células humanas expresan tres IDH diferentes: IDH1, IDH2 e IDH3.
La IDH1 es citosólica NADP dependiente, la IDH2 es mitocondrial NADP dependiente, muy parecida estructuralmente a la IDH1 y, por último, la IDH3 utiliza NAD como cofactor en la mitocondria.[2] [3] [4]
Este estudio se centró en la isoforma IDH1 debido a que se encontró más evidencia al respecto y encajó mejor con el objetivo de añadir escenas 3D que permitieran enriquecer los artículos consultados.
Estructura de la IDH1 y sitio activo
La IDH1, con código1T09 en PDB, tiene como ligando la NADP y por lo tanto forma un complejo binario IDH1-NADP. Existe otra IDH1 con código 1T0L en PDB, que conforma una estructura cuaternaria ya que contiene tres ligandos: ICT, NADP y Ca2+, por lo que el complejo cuaternario será IDH1- ICT-NADP-Ca2+. Su estructura contiene cuatro subunidades: A, B, C y D, cada una con sus tres ligandos. Sin importar el número de subunidades que presente cada una de las estructuras de IDH1 (1T09 complejo binario y 1T0L complejo cuaternario), las dos estructuras presentan tres dominios:
- Dominio grande (L): residuos 1-103 y 286-414.
- Dominio pequeño (S): residuos 104-136 y 186-285.
- Dominio clasp (C): residuos 137-185.
Además, presentan un segmento regulatorio formado por los residuos 271-286 en el sitio activo, a partir de aquí llamado péptido regulador.
Escena 1. Representación de los dominios de la enzima ID09
La conformación de la subunidad A (rojo) en el complejo binario (IDH-NADP) se parece más a la forma abierta. Proponen que la conformación abierta de IDH puede representar a la forma inactiva de la enzima. La conformación de IDH de la subunidad B (azul) del complejo binario (IDH-NADP) está entre la forma abierta y la cerrada. Proponen que la forma semiabierta de la IDH puede representar una forma intermedia entre la conformación activa y la inactiva.
Escena 0. Conservación evolutiva"
Escena 2. Superposición de las subunidades A y B de IT09
Con esta escena se quiso observar el cambio conformacional de abierta a semiabierta.
Se hizo un alineamiento jFATCAT flexible y dio como resultado un RMSD de 0.95 angstroms, lo cual indicó que había muy pocas diferencias entre una subunidad y la otra.
Lo interesante de esta escena es que permitió ver el cambio conformacional que se hablaba anteriormente de abierta (cadena A) a semiabierta (cadena B).
Escena 3. Superposición de las cadenas A de 1T09 y 1T0L El objetivo de esta escena fue poder observar el cambio conformacional del péptido regulador cuando entran los ligandos ICT y Ca2+ en el complejo cuaternario. Se hizo un alineamiento entre las cadenas A de 1T09 y 1T0L jFATCAT flexible, lo cual resultó en un RMSD de 3.34 Angstroms, valor que indica ciertos cambios en las estructuras que observamos en la escena. La imagen de la izquierda (activa) es la cadena A de la 1T0L (cuaternario) con ICT en blanco y Ca2+ en cyan, A la derecha (inactiva) la cadena A de la 1T09 (binario). NADP amarillo y péptido regulatorio en naranja en ambas imágenes. Los colores de los dominios se mantienen de acuerdo a las anteriores escenas.
Además, el modelo de trabajo de Xiang Xu et al. postula que la IDH puede utilizar el segmento regulatorio y las interacciones de Asp279 (morado) con Ser94 (verde pistacho) para inactivar la enzima, lo cual se comprueba con la escena 3D puesto que se observó que al entrar ICT y Ca2+, la proteína reguladora cambia de conformación al igual que lo hacen Asp279 y Ser94.
La unión competitiva del isocitrato induce el replegamiento de la proteína reguladora y los cambios conformacionales generales y consecuentemente para activar la enzima.
Escena 4. Alineamiento 1T09 con 1T0L, cadenas A
Con el siguiente comando: contact {ICT} full connect se buscaron los contactos del ICT en Jmol, y como se esperaba, aparecieron los 15 contactos que se describe en el paper:
- Thr77, Ser94, Arg100, Arg109, Arg132, Tyr139, y Asp275 en una subunidad
- Lys212, Thr214, y Asp252 en la subunidad adyacente
- 3 moléculas de agua, NADP y Ca2+
Escena 5. Interacción Arg132 con ICT
Mutación en la Arg132 y su relación con el cáncer
Recientemente se ha descubierto que el NADP-IDH citosólico humano está implicado en la tumorigénesis. En particular, las mutaciones IDH1 derivadas de tumores identificadas ocurren principalmente en Arg132, y la mutación R132H es la más prevalente.
Arg132 desempeña múltiples funciones funcionales en la reacción catalítica; en particular, la mutación R132H dificulta los cambios conformacionales desde el estado inicial de unión a las ICT al estado previo a la transición, lo que lleva al deterioro de la actividad IDH.
Las mutaciones en la enzima causan la aparición de una nueva actividad catalítica que provoca la conversión de alfa-cetoglutarato (α-KG) en 2-hidroxiglutarato (2-HG), que desregula un conjunto de dioxigenasas dependientes de α-KG. Específicamente, debido a la estructura similar entre 2-HG y α-KG, 2-HG bloquea competitivamente la unión de α-KG a histonas desmetilasas, como la desmetilasa específica de lisina (KDM), y luego aumenta la metilación de histonas. El 2-HG también inhibe la actividad de diez a once hidroxilasas de translocación, que son importantes para la metilación global del ADN.
Por otro lado, los altos niveles de 2-HG debido a las mutaciones de IDH, estabilizan el factor HIF alfa debido a que disminuyen los niveles de una endostatina de HIF-1α lo cual resulta en la upregulación de los genes dependientes de la endostatina HIF-1α, que incluyen el VEGF factor de crecimiento vascular endotelial.
Escena 6.
Distintas conformaciones pueden representar diferentes estados enzimáticos
Los dos complejos (binario y cuaternario) contienen tres conformaciones diferentes. La conformación general del complejo cuaternario se parece a la cerrada. La zona activa puede adoptar una conformación compacta o cerrada.
En la evidencia, sugieren que las diferencias conformacionales de los segmento regulatorio en los tres conformadores de IDH son probablemente relacionado con las funciones biológicas de la enzima, en lugar de un artefacto, y que las distintas conformaciones de IDH observadas en las dos estructuras complejas probablemente sean biológicamente relevantes y puede representar diferentes estados enzimáticos.
Conclusión
Se ha comprobado la gran utilidad de las representaciones en 3D para biomoléculas complejas en publicaciones científicas. La visualización en 3D permite acercarse a las estructuras para así entender mejor su composición y función, así como las uniones con sus ligandos y sus cambios conformacionales; cosas que en el mundo 2D a menudo se escapan.
Your Heading Here (maybe something like 'Structure')
| |||||||||||
References
- ↑ Protein Data Bank in Europe (PDBe). (n.d.).
- ↑ Yang, B., Zhong, C., Peng, Y. et al. Molecular mechanisms of “off-on switch” of activities of human IDH1 by tumor-associated mutation R132H. Cell Res 20, 1188–1200 (2010). https://doi.org/10.1038/cr.2010.145
- ↑ Xiang Xu, Jingyue Zhao, Zhen Xu, Baozhen Peng, Qiuhua Huang, Eddy Arnold, Jianping Ding, Structures of Human Cytosolic NADP-dependent Isocitrate Dehydrogenase Reveal a Novel Self-regulatory Mechanism of Activity*, Journal of Biological Chemistry, Volume 279, Issue 32, 2004, Pages 33946-33957, ISSN 0021-9258, https://doi.org/10.1074/jbc.M404298200
- ↑ Chen X, Yang P, Qiao Y, Ye F, Wang Z, Xu M, Han X, Song L, Wu Y, Ou WB. Effects of cancer-associated point mutations on the structure, function, and stability of isocitrate dehydrogenase 2. Sci Rep. 2022 Nov 5;12(1):18830. doi: 10.1038/s41598-022-23659-y. PMID: 36335201; PMCID: PMC9637083.