1quf (Italian)
From Proteopedia
Contents |
This is a placeholder
This is a placeholder text to help you get started in placing a Jmol applet on your page. At any time, click "Show Preview" at the bottom of this page to see how it goes.
Replace the PDB id (use lowercase!) after the STRUCTURE_ and after PDB= to load and display another structure.
| |||||||
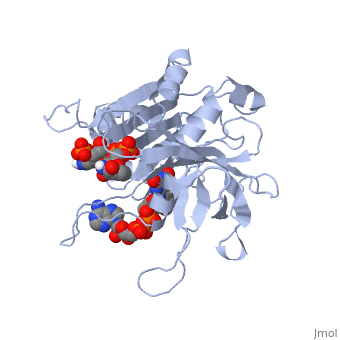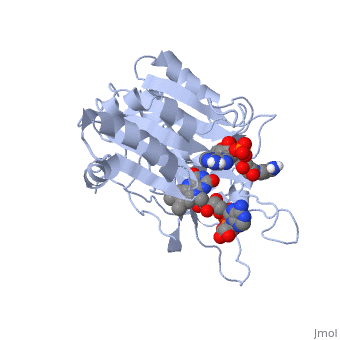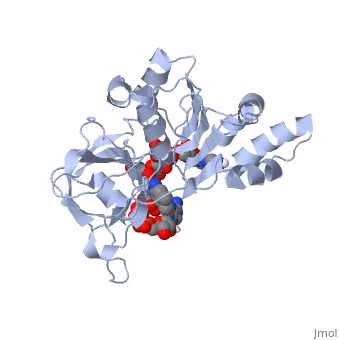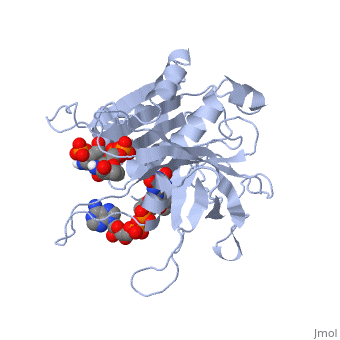
| 1quf, resolution 2.25Å () | |||||||
|---|---|---|---|---|---|---|---|
| Ligands: | , | ||||||
| Activity: | Ferredoxin--NADP(+) reductase, with EC number 1.18.1.2 | ||||||
| |||||||
| |||||||
| Resources: | FirstGlance, OCA, RCSB, PDBsum | ||||||
| Coordinates: | save as pdb, mmCIF, xml | ||||||
STRUTTURA AI RAGGI X DELLA FERREDOXIN:NADP+ REDUCTASE DAL CIANOBATTERIO ANABAENA PCC 7119 A 1.8 ANGSTROM
La struttura nel cristallo della ferredoxin:NADP+ reductase (FNR) dal cianobatterio Anabaena PCC 7119 è stata determinata a una risoluzione di 2.6 A con sostituzione isomorfa multipla ed affinata usando dati tra 15.0 a 1.8 A, raccolti a 4 gradi C, fino ad un fattore R di 0.172. Il modello include 303 residui, il cofattore flavin adenine dinucleotide (FAD), un ione solfato localizzato al luogo di legame del NADP+ e 328 molecole di acqua. La struttura di Anabaena FNR, incluso il FAD, una rete di molecole di acqua intrinseche ed una grande cavità idrofobica nel dominio C-terminale, assomiglia a quella dell'enzima di spinacio. Le differenze notevoli concernono l'alfa-elica supplementare corta (residui 172 a 177 in Anabaena FNR), e residui Arg 100 ed Arg 233 i quali legano il NADP+ invece di Lys 116 e Lys 244 nell'enzima di spinacio. Cristalli di un complesso di Anabaena FNR con NADP+ furono ottenuti. Il modello del complesso è stato affinato fino a un fattore R di 0.186, usando dati di diffrazione ai ragi X da 15 fino a 2.25 A di risoluzione raccolti a -170 gradi C. Questo modello include 295 residui, il FAD, il NADP+ intero (con un'occupazione di 0.8) e 444 molecole di acqua. La parte 2'-5' adenine del NADP+ si lega alla proteina come il 2'-fosfo-5'-AMP alla FNR di spinacio. La parte nicotinamide è girata verso la superficie della proteina invece di accatastarsi sopra l'annello isoalloxazine del FNR, come sarebbe richiesto per il trasferimento di idride. Il modello del complesso è in accordo con studi biochimichi precedenti, visto che i residui Arg 100 ed Arg 233 sono implicati nel collegamento del NADP+, e i residui Arg77, Lys 53 e Lys 294 situati sul lato FAD dell'enzima, rimangono liberi per interagire con ferredossina e flavodossina, i partner fisiologici della ferredoxin:NADP reductase.
Riassunto tradotto da MEDLINE®/PubMed®, una banca dati del U.S. National Library of Medicine.
A proposito di questa Struttura
1QUF è una struttura a 1 catena di sequenza di Anabaena sp.. Dalle informazioni cristallografiche completi sono disponibili su OCA.
Riferimento
- Serre L, Vellieux FM, Medina M, Gomez-Moreno C, Fontecilla-Camps JC, Frey M. X-ray structure of the ferredoxin:NADP+ reductase from the cyanobacterium Anabaena PCC 7119 at 1.8 A resolution, and crystallographic studies of NADP+ binding at 2.25 A resolution. J Mol Biol. 1996 Oct 18;263(1):20-39. PMID:8890910 doi:10.1006/jmbi.1996.0553
Page seeded by OCA on Tue Feb 17 04:01:53 2009
Categories: Anabaena sp. | Frey, M. | Serre, L. | Vellieux, F M.D. | Fad | Flavoprotein | Fnr | Hycobilisome | Nadp | Nadp+ reductase | Oxidoreductase | Thylakoid membrane