2x0s (French)
From Proteopedia
| |||||||||
| 2x0s, resolution 3.00Å () | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Activity: | Pyruvate, phosphate dikinase, with EC number 2.7.9.1 | ||||||||
| |||||||||
| |||||||||
| Resources: | FirstGlance, OCA, PDBsum, RCSB | ||||||||
| Coordinates: | save as pdb, mmCIF, xml | ||||||||
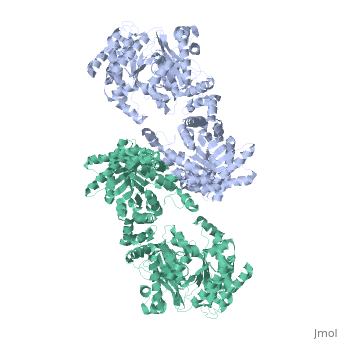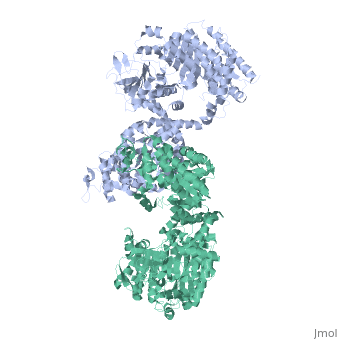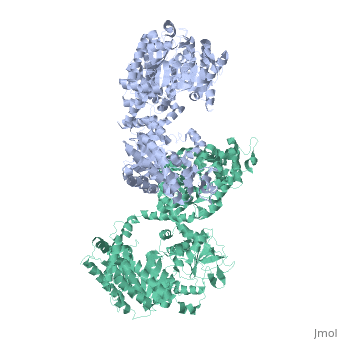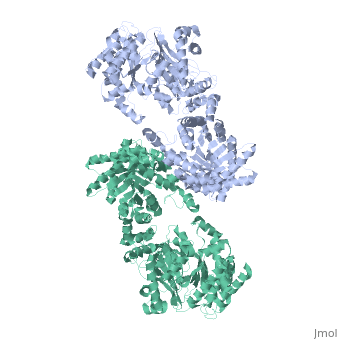
STRUCTURE CRISTALLOGRAPHIQUE A 3.0 A DE RÉSOLUTION DE LA PYRUVATE PHOSPHATE DIKINASE GLYCOSOMALE DE TRYPANOSOMA BRUCEI
La structure cristallographique de l'enzyme glycosomale pyruvate phosphate dikinase du parasite protozoaire africain Trypanosoma brucei a été résolue à 3.0 A de résolution par remplacement moléculaire. Le modèle de recherche était la structure à 2.3 A de résolution de l'enzyme de Clostridium symbiosum. En raison des orientations relatives différentes des domaines et des sous-domaines dans les deux structures, le remplacement moléculaire n'a pu être réalisé qu' en positionnant ces éléments (quatre corps en tout) séquentiellement dans l'unité asymétrique du cristal de groupe d'espace P2(1)2(1)2, qui contient une sous-unité de phosphate pyruvate dikinase (PPDK). Le modèle affiné, comprenant 898 résidus et 188 molécules de solvant par sous-unité, a un résiduel crystallographique Rf = 0.245 (facteur R libre Rfree = 0.291) et montre une stéreochimie satisfaisante. Huit régions, comprenant un total de 69 résidus à la surface de la molécule, sont désordonnées dans cette forme cristalline. Les sous-unités de PPDK sont arrangées autour de l'axe cristallographique d'ordre comme un dimère, analogue à celui observé dans l'enzyme de C. symbiosum. La comparaison des deux structures a été effectuée par superposition des deux modèles. Bien que le repliement de chaque domaine ou sous-domaine soit semblable, les orientations relatives de ces éléments constitutifs sont différentes dans les deux structures. L'enzyme trypanosomale est plus "recourbée" que l'enzyme bactérienne, avec la courbure s'accroissant depuis le centre de la molécule (proche de l'axe d'ordre 2 moléculaire) vers la périphérie où le domaine N-terminal est situé. En conséquence de cette courbure accrue et des différentes positions relatives des sous-domaines, la fente de liaison du nucléotide dans le domaine amino-terminal est plus large dans la PPDK de T. brucei: le fragment N-terminal du domaine amino-terminal est situé à distance de l'histidine catalytique 482, compétente pour la réaction de phospho-transfert (à peu près à 10 A de distance). Nos observations suggèrent que les exigences de mouvements de domaines pendant la catalyse par l'enzyme pourraient inclure l'élargissement de la fente de liaison du nucléotide, pour permettre l'accès et le départ des ligands AMP ou ATP.
The 3.0 A resolution crystal structure of glycosomal pyruvate phosphate dikinase from Trypanosoma brucei., Cosenza LW, Bringaud F, Baltz T, Vellieux FM, J Mol Biol. 2002 May 17;318(5):1417-32. PMID:12083528
From MEDLINE®/PubMed®, a database of the U.S. National Library of Medicine.
Résumé traduit de MEDLINE®/PubMed®, une base de données de la U.S. National Library of Medicine.
Au sujet de cette structure
2X0S est une structure à 1 chaîne de séquence de Trypanosoma brucei. Des informations cristallographiques complètes sont disponibles sur OCA. Numéros biologiques appropriés pour cette protéine sur B10NUMB3R5
Références
- Cosenza LW, Bringaud F, Baltz T, Vellieux FM. The 3.0 A resolution crystal structure of glycosomal pyruvate phosphate dikinase from Trypanosoma brucei. J Mol Biol. 2002 May 17;318(5):1417-32. PMID:12083528
- Cosenza LW, Bringaud F, Baltz T, Vellieux FM. Crystallization and preliminary crystallographic investigation of glycosomal pyruvate phosphate dikinase from Trypanosoma brucei. Acta Crystallogr D Biol Crystallogr. 2000 Dec;56(Pt 12):1688-90. PMID:11092947
- Bringaud F, Baltz D, Baltz T. Functional and molecular characterization of a glycosomal PPi-dependent enzyme in trypanosomatids: pyruvate, phosphate dikinase. Proc Natl Acad Sci U S A. 1998 Jul 7;95(14):7963-8. PMID:9653123