User:Nathália Gagliardi Saldys/Sandbox 1
From Proteopedia
| Line 24: | Line 24: | ||
Ela é uma HDAC de classe IIb, enzimas dependentes de íons Zn2+, que são essenciais para a catálise: o Zn²⁺ polariza a carbonila do grupo acetilado, facilitando seu ataque por uma molécula de água ativada (ataque nucleofílico), o que remove o grupo acetil da lisina. | Ela é uma HDAC de classe IIb, enzimas dependentes de íons Zn2+, que são essenciais para a catálise: o Zn²⁺ polariza a carbonila do grupo acetilado, facilitando seu ataque por uma molécula de água ativada (ataque nucleofílico), o que remove o grupo acetil da lisina. | ||
| - | Estudos com estruturas cristalinas de raios X de HDAC6 <scene name='10/1082407/Cd2_homo_sapiens/1'>CD2 de ''H.sapiens'' (Humano)</scene> e HDAC6 CD1 e CD2 de | + | Estudos com estruturas cristalinas de raios X de HDAC6 <scene name='10/1082407/Cd2_homo_sapiens/1'>CD2 de ''H.sapiens'' (Humano)</scene> e HDAC6 CD1 e <scene name='10/1082407/Cd2/2'>CD2 de D.rerio (peixe-zebra)</scene>, revelaram que a enzima do peixe-zebra possui uma conservação estrutural significativa e pode ser usada como substituto funcional da enzima humana para análises estruturais. |
Esses mesmos estudos mostram que o domínio CD2 da HDAC6 humana apresenta um íon Zn²⁺ coordenado diretamente por '''Asp612, His614 e Asp705''', além de uma molécula de água, que interage por pontes de hidrogênio com '''His573 e His574''', formando parte do sítio catalítico. | Esses mesmos estudos mostram que o domínio CD2 da HDAC6 humana apresenta um íon Zn²⁺ coordenado diretamente por '''Asp612, His614 e Asp705''', além de uma molécula de água, que interage por pontes de hidrogênio com '''His573 e His574''', formando parte do sítio catalítico. | ||
Revision as of 17:08, 8 July 2025
Contents |
HDAC6 - Estrutura e função
Sobre a proteína e suas funções
|
A HDAC 6 é uma proteína citoplasmática da família HDAC e tem como principal função a desacetilação de uma ampla gama de substratos, com destaque para proteínas não histônicas. Entre os substratos conhecidos estão α-tubulina, cortactina, Hsp90, peroxirredoxina e Tat.
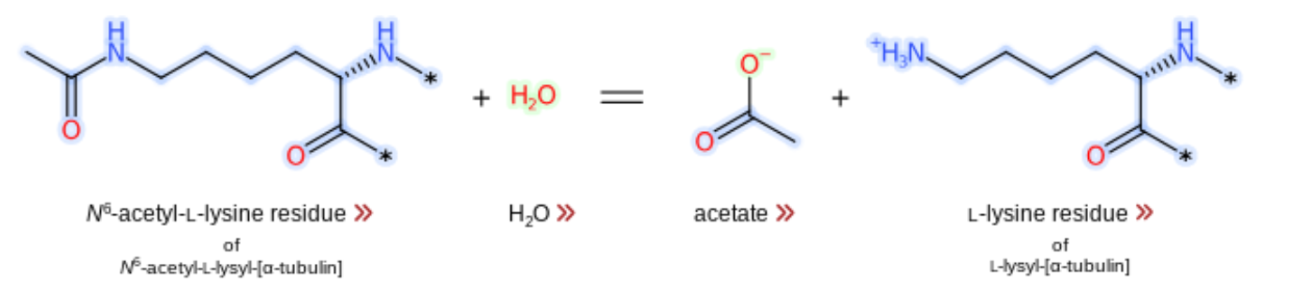
A HDAC 6 pode se associar aos microtúbulos e desacetilar a proteína α-tubulina catalisando a remoção do grupo acetil (Figura 1). Essa desacetilação mediada por HDAC6 regula a motilidade celular dependente de microtúbulos. Além disso, essa acetilação reversível regula importantes processos biológicos como metabolismo de histonas, transcrição gênica e desmontagem dos cílios, importante para a regulação da homeostase ciliar.
E além dessa função de desacetilação, essa proteína desempenha um papel fundamental na degradação de proteínas mal dobradas: quando proteínas mal dobradas são abundantes demais para serem degradadas pelo sistema de redobramento de chaperonas e pelo ubiquitina-proteassoma, a HDAC6 auxilia na sua eliminação por vias alternativas, como a autofagia. Além disso, a HDAC6 está envolvida na regulação epigenética associada à progressão de alguns tipos de câncer.
Perante essas diversas funções é de enorme importância conhecer melhor a estrutura dessa proteína e correlacionar cada domínio catalítico com alguma de suas funções, até para desenvolvimento de possíveis fármacos.
Figura 1. Desacetilação da α-tubulina
Estrutura da proteína relacionada à função catalítica (desacetilação)
A HDAC 6 é única dentro da família das histonas desacetilases por apresentar um domínio catalítico duplo ( e ). Ambos são capazes de realizar a função de desacetilação, mas o CD2 (domínio catalítico 2) é o principal responsável pela desacetilação de proteínas não histônicas, como a α-tubulina.
A HDAC6 catalisa a desacetilação de K40 (Lisina 40) na subunidade α-tubulina do microtúbulo, regulando assim a dinâmica dos microtúbulos: a inibição do HDAC6 leva à hiperacetilação da α-tubulina e à supressão da dinâmica dos microtúbulos, enquanto a superexpressão do HDAC6 reduz a acetilação da tubulina e aumenta a motilidade celular.
Ela é uma HDAC de classe IIb, enzimas dependentes de íons Zn2+, que são essenciais para a catálise: o Zn²⁺ polariza a carbonila do grupo acetilado, facilitando seu ataque por uma molécula de água ativada (ataque nucleofílico), o que remove o grupo acetil da lisina.
Estudos com estruturas cristalinas de raios X de HDAC6 e HDAC6 CD1 e , revelaram que a enzima do peixe-zebra possui uma conservação estrutural significativa e pode ser usada como substituto funcional da enzima humana para análises estruturais.
Esses mesmos estudos mostram que o domínio CD2 da HDAC6 humana apresenta um íon Zn²⁺ coordenado diretamente por Asp612, His614 e Asp705, além de uma molécula de água, que interage por pontes de hidrogênio com His573 e His574, formando parte do sítio catalítico.
Mecanismo Catalítico
As hidrolases de zinco ativam um grupo carbonila do substrato para ataque nucleofílico direto por uma molécula de água usando coordenação Zn2 + e uma interação de ligação de hidrogênio para polarizar o grupo carbonila; a molécula de água nucleofílica é ativada pela coordenação Zn2 + e uma base geral. Logo, a carbonila do grupo acetilado na lisina é polarizada por interações com o Zn²⁺ e uma base geral, o que facilita o ataque nucleofílico por uma molécula de água ativada — mecanismo típico de hidrolases de zinco.
Em resumo, o objetivo de uma desacetilase é remover o grupo acetil (–COCH₃) de uma lisina acetilada (Ac-Lys) presente em proteínas como α-tubulina. Na primeira etapa desse processo a lisina acetilada do substrato é encaixada no domínio CD2 e o grupo acetil se aproxima do centro catalítico contendo o íon Zn²⁺. Em seguida, o grupo carbonila (C=O) do grupo acetil interage com o íon Zn²⁺, polarizando o carbono carbonílico (o tornando mais eletrofílico). Ao mesmo tempo, um resíduo de base geral desprotona uma molécula de H₂O, gerando um íon hidroxila (OH⁻) reativo, o qual ataca o carbono da carbonila do grupo acetil, formando um intermediário tetraédrico. Esse intermediário colapsa e libera um acetato (CH₃COO⁻), assim, a lisina fica desacetilada e é liberada do sítio ativo.
Referências
UNIPROT. Q9UBN7 – Histone deacetylase 6 (HDAC6) [Homo sapiens]. Disponível em: https://www.uniprot.org/uniprotkb/Q9UBN7/entry. Acesso em: 10 jun. 2025.
EUROPEAN BIOINFORMATICS INSTITUTE. AlphaFold Protein Structure Database – HDAC6 (Q9UBN7). Disponível em: https://alphafold.ebi.ac.uk/entry/Q9UBN7. Acesso em: 10 jun. 2025.
HUBBERT, C.; GUARDIOLA, A.; SHAO, R. et al. HDAC6 is a microtubule-associated deacetylase. Nature, [S.l.], v. 417, n. 6887, p. 455–458, 2002. DOI: 10.1038/417455a. Disponível em: https://pubmed.ncbi.nlm.nih.gov/12024216/. Acesso em: 10 jun. 2025.
RAN, J. et al. Deacetylation of α-tubulin and cortactin is required for HDAC6 to trigger ciliary disassembly. Scientific Reports, [S.l.], v. 5, p. 12917, 2015. DOI: 10.1038/srep12917. Disponível em: https://pubmed.ncbi.nlm.nih.gov/26246421/. Acesso em: 10 jun. 2025.
HAI, Y.; CHRISTIANSON, D. W. Histone deacetylase 6 structure and molecular basis of catalysis and inhibition. Nature Chemical Biology, [S.l.], v. 12, n. 9, p. 741–747, 2016. DOI: 10.1038/nchembio.2134. Disponível em: https://www.nature.com/articles/nchembio.2134. Acesso em: 10 jun. 2025.
This is a sample scene created with SAT to by Group, and another to make of the protein. You can make your own scenes on SAT starting from scratch or loading and editing one of these sample scenes.
</StructureSection>